
Lille, France. Bioinformatics, data-structures for DNA/RNA.

www.nature.com/articles/d41...
You can find it at gitlab (gitlab.com/rahmannlab/c...) or install it via PyPI or Bioconda.

You can find it at gitlab (gitlab.com/rahmannlab/c...) or install it via PyPI or Bioconda.
1/
1/

Vous êtes personnel de l’éducation nationale, confronté aux conséquences de l’adhésion obligatoire à la complémentaire santé MGEN : témoignez en remplissant ce bref formulaire afin que je puisse interpeler le ministre de l'Éducation nationale.
Vous êtes personnel de l’éducation nationale, confronté aux conséquences de l’adhésion obligatoire à la complémentaire santé MGEN : témoignez en remplissant ce bref formulaire afin que je puisse interpeler le ministre de l'Éducation nationale.

AI models still lag far behind the level of protein prediction. The best scores rely on old-school templates. Human intuition still matters. 7/8
AI models still lag far behind the level of protein prediction. The best scores rely on old-school templates. Human intuition still matters. 7/8
blog.genesmindsmachines.com/p/python-is-...
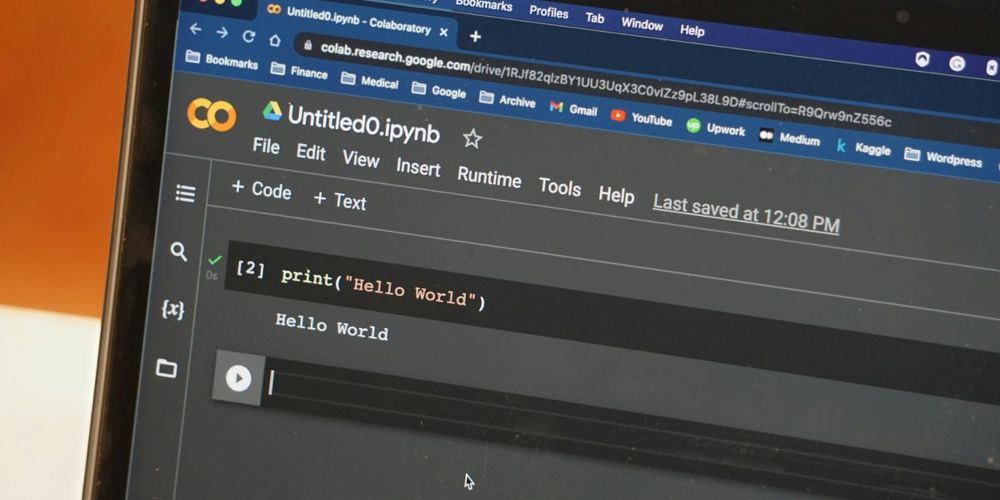
blog.genesmindsmachines.com/p/python-is-...
La question de la sécurité doit être au coeur des developpements, y compris en bioinformatique. Au dela du cas d'ONT, cela pose la question des formations ciblées "devs scientifique"et des moyens.
La question de la sécurité doit être au coeur des developpements, y compris en bioinformatique. Au dela du cas d'ONT, cela pose la question des formations ciblées "devs scientifique"et des moyens.
Bravo aux organisateurs et intervenants !
carep-paris.org/annonce/comm...

Bravo aux organisateurs et intervenants !
carep-paris.org/annonce/comm...
I'm looking for studies where both Illumina and ONT sequencing were performed on the same samples from soil, human, ruminent, and other sample types for comparison. Bonus if those studies include PacBio data.
Please help and share!
I'm looking for studies where both Illumina and ONT sequencing were performed on the same samples from soil, human, ruminent, and other sample types for comparison. Bonus if those studies include PacBio data.
Please help and share!

1) regarder des flims dans le train
2) lire des PDF
3) prendre des notes manuscrites (typiquement pour le boulot)
1) regarder des flims dans le train
2) lire des PDF
3) prendre des notes manuscrites (typiquement pour le boulot)

Franchement ça me fait tout bizarre d'être d'accord avec France Universités. C'est dire si les pressions de notre ministre (et la décision du CDF) sont inquiétantes.
franceuniversites.fr/actualite/fr...
#LibertéAcadémique

Franchement ça me fait tout bizarre d'être d'accord avec France Universités. C'est dire si les pressions de notre ministre (et la décision du CDF) sont inquiétantes.

The first talk, by Irene Kaplow, focused on Challenges in Predicting Enhancer Activity Differences Between Species
doi.org/10.1186/s12864-022-08450-7


![OCR Ortholog Open Chromatin Status Prediction Framework Overview. a We trained a convolutional neural network (CNN) for predicting brain open chromatin using sequences underlying brain open chromatin region (OCR) orthologs in a small number of species and used the CNN to predict brain OCR ortholog open chromatin status across the species in the Zoonomia Consortium. Specifically, we used the sequences underlying the orthologs for which we have brain open chromatin data to train a CNN for predicting open chromatin. Then, we used the CNN to predict the probability of brain open chromatin for all brain OCR orthologs; predictions are illustrated on the right. Animals for which we do not have open chromatin data are in dark gray instead of black to indicate that their brain open chromatin is imputed. While we cannot evaluate the accuracy of most of our predictions, obtaining open chromatin data from most tissues in most species is infeasible, so predictions might be the best OCR annotations that we can obtain. b To demonstrate that our models can accurately predict whether sequence differences between species are associated with open chromatin differences, in addition to the evaluations described in previous work [57], we evaluated our performance on species-specific open chromatin for a species not used in model training and clade-specific open and closed chromatin for clades not used in model training. Since such regions often comprise a minority of OCR orthologs, models could obtain good overall performance while obtaining poor performance on such regions. We also evaluated our performance on tissue-specific open and closed chromatin for a tissue not used in model training, where we expect models to predict 0 if model learns sequence signatures related to the tissue used in training. c Full mouse test set and lineage-specific OCR accuracy evaluations for mouse sequence-only brain model, illustrating that, even for the best of these models,](https://cdn.bsky.app/img/feed_thumbnail/plain/did:plc:jkd2l647lviezhenqed454lh/bafkreicgkhegfuippjqhdt2tkku6dr74stuf5znohmtovfk5zojt5w7xkq@jpeg)
The first talk, by Irene Kaplow, focused on Challenges in Predicting Enhancer Activity Differences Between Species
doi.org/10.1186/s12864-022-08450-7


