Olivier Namy 🧬🧪
@onamy.bsky.social
1.3K followers
270 following
94 posts
Phd, director of research, RNA Biology & Ribosome, former president Section 21 #CNRS, #RNA and #ribosome fan, specialist in translation regulations at #I2BC
ORCID: 0000-0002-1143-5961
Posts
Media
Videos
Starter Packs
Pinned
Olivier Namy 🧬🧪
@onamy.bsky.social
· Nov 12
Reposted by Olivier Namy 🧬🧪
Reposted by Olivier Namy 🧬🧪
cryoEM papers
@cryoempapers.bsky.social
· Aug 12
Reposted by Olivier Namy 🧬🧪
Reposted by Olivier Namy 🧬🧪
Reposted by Olivier Namy 🧬🧪
Reposted by Olivier Namy 🧬🧪
Reposted by Olivier Namy 🧬🧪
Molecular Cell
@cp-molcell.bsky.social
· May 17
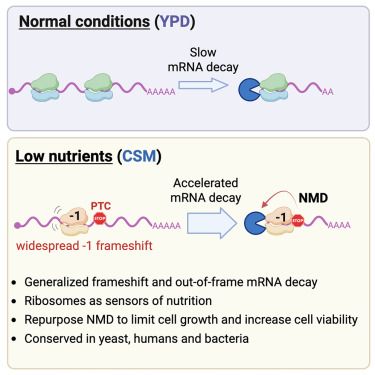
Ribosomes modulate transcriptome abundance via generalized frameshift and out-of-frame mRNA decay
Zhang et al. identify a regulatory mechanism for how cells adapt to nutrient scarcity through widespread −1 ribosomal frameshifts, culminating in accelerated mRNA decay. This process, dependent on codon optimality and conserved across species, establishes direct feedback coupling the translation of new proteins with the stability of the mRNA that encodes for them.
dlvr.it
Reposted by Olivier Namy 🧬🧪
Olivier Namy 🧬🧪
@onamy.bsky.social
· Apr 6
Olivier Namy 🧬🧪
@onamy.bsky.social
· Apr 6